Trending Now
+(34) 669 817 249 / +(34) 876 643 011


http://winepi.net/covid19.htm
Uno de los desafíos que afrontan los microbiólogos es poner un nombre adecuado a los virus y las bacterias que presentan diferencias genéticas relevantes dentro de una misma especie, pero sin que lleguen a ser especies diferentes. SARS-CoV-2 es un buen modelo ya que desde el principio mostró cambios genéticos importantes (mutaciones y recombinaciones) que obligaron a plantear distintas nomenclaturas. El problema aparece cuando no existe un consenso sobre el sistema a seguir.
Para SARS-CoV-2 existen tres propuestas principales y para entenderlas lo mejor es poner un ejemplo con una de las variantes. Empezaremos por Phylogenetic Assignment of Named Global Outbreak Lineages, que abreviado en inglés sería PANGOLIN, haciendo un guiño al papel de este animal como presunto reservorio intermedio entre el murciélago y los humanos en el salto zoonótico del virus. Estos linajes PANGO usan una combinación de letras y números para hacer el seguimiento de la evolución de las distintas variantes y subvariantes, como por ejemplo la B.1.1.7 (luego pondré más ejemplos).
Otro de los sistemas para denominar las variantes víricas procede de Nextstrain (https://nextstrain.org/ncov/) que permite hacer un seguimiento casi en tiempo real de la clasificación filogenética del virus y utiliza una nomenclatura mucho más simple basada en un número correspondiente al año de inicio del clado (simplificando podríamos decir que sería un sinónimo de los linajes PANGO) y una letra asignada de forma correlativa, de manera que el linaje PANGO B.1.1.7 se corresponde con el clado Nextstrain 20I.
Cuando se detecta en algún país una variante emergente con diferencias relevantes en su genoma se clasifica como VOI y si se confirma su potencial para propagarse globalmente pasa a la categoría VOC
Sin embargo, esta doble nomenclatura es confusa incluso para los virólogos más expertos, así que la Organización Mundial de la Salud (OMS) y el Grupo Interagencial del SARS-CoV-2 (SIG) del Gobierno de EE.UU. decidieron clasificar las variantes con importancia epidemiológica y sanitaria en diferentes categorías: VOI (variante de interés), VOC (variante de preocupación), VUM (variante bajo monitoreo) y VOHC (variante de gran consecuencia).
Cuando se detecta en algún país una variante emergente con diferencias relevantes en su genoma se clasifica como VOI y si se confirma su potencial para propagarse globalmente pasa a la categoría VOC. Una vez que una variante VOC se convierte en residual pasa a ser VUM (en el caso de EE.UU.), mientras que la OMS la clasifica como VOC/VOI previamente circulantes. Y la última categoría (VOHC) se reserva para aquellas variantes catastróficas de las cuales no hemos tenido ninguna hasta al momento (y que siga así). A aquellas variantes que se clasificaban como VOI la OMS les asignaba una letra del alfabeto griego, y B.1.1.7, alias 20I, fue la primera de ellas y se nombró como Alfa.
Actualmente sólo Omicron se mantiene como VOC, y las anteriores Alfa, Beta, Gamma, Delta, Epsilon, Eta, Zeta, Iota, Kappa, Lambda y Mu han pasado a la categoría VUM como algo ya superado. Notarán que faltan Nu y Xi entre Mu y Omicron, ya que se excluyeron porque Nu se pronunciaba de forma similar a New (nuevo en inglés) y Xi por ser un apellido frecuente en China. Si les interesa las siguientes variantes de la OMS serían Pi, Ro, Sigma, Tau, Ipsilon, Phi, Chi, Psi y Omega, y la intención era continuar con nombres de constelaciones.
Sin embargo, nos hemos “atascado” en Omicron, y eso no indica que no esté evolucionando ya que incluye numerosos clados Nextstrain (21K, 21L, 22A, 22B, 22C, 22D, 22E y 22F) que a su vez corresponde con cinco linajes PANGO (BA.1, BA.2, BA.3, BA.4 y BA.5) con numerosos sublinajes. Así que dejaremos para una próxima colaboración la nomenclatura de Omicron.
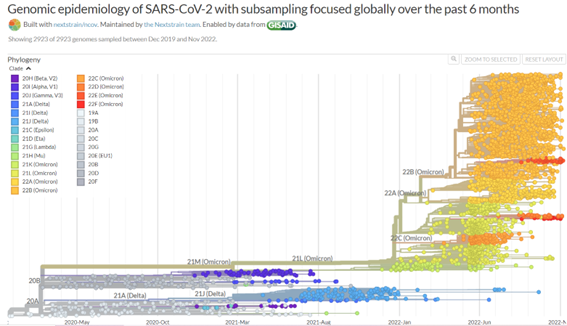
Este es el árbol filogenético que genera Nextstrain. Se accede directamente a él desde el enlace que encontraréis en el texto. En este ejemplo se usan los clados Nextstrain con su equivalencia OMS.
+(34) 669 817 249 / +(34) 876 643 011